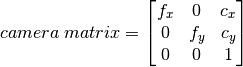이 글의 원문은 https://opencv-python-tutroals.readthedocs.io/en/latest/py_tutorials/py_calib3d/py_pose/py_pose.html 입니다.
이전 글은 카메라 보정에 대한 내용으로, 카메라 메트릭스와 왜곡 계수 등을 구했습니다. 주어진 패턴 이미지를 통해 우리는 이미지 안의 패턴의 자세를 계산하기 위한 정보를 이용할 수 있고 객체가 공간상에 어떻게 놓여있는지 파악할 수 있습니다. 평면 객체에 대해서는, Z값을 0으로 가정한다면.. 이미지의 이러한 객체에 대한 자세 문제는 카메라가 공간 상에 어떻게 위치해 있는지의 문제가 됩니다. 그래서, 만약 공간 상에 객체가 어떻게 놓여 있는지를 안다면, 3차원 효과를 시뮬레이션하기 위해 이미지 상에 2차원 도형을 그려 넣을 수 있습니다.
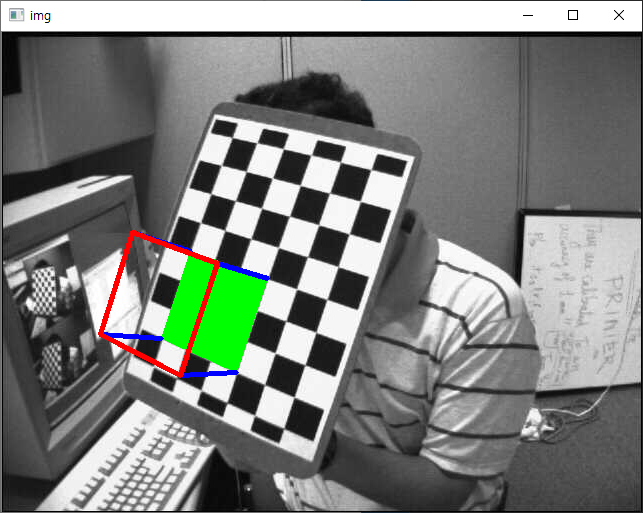
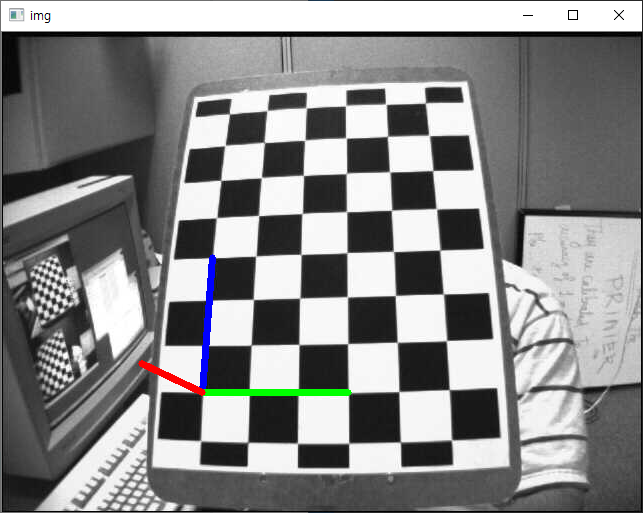
우리의 목표는 체스판의 첫번째 코너 지점 위에 3차원 좌표축(X, Y, Z 축)을 그려 넣는 것입니다. X 측은 파랑색으로, Y축은 초록색으로 Z축은 빨간색으로 그려 봅시다. 그래서 효과면에서 볼 때, Z축은 체스판 상에 수직으로 느껴져야 합니다.
가장 먼저, 이전 카메라 보정 결과로부터 카메라 행렬과 왜곡계수를 계산해 봅시다.
import numpy as np
import cv2
import glob
criteria = (cv2.TERM_CRITERIA_EPS + cv2.TERM_CRITERIA_MAX_ITER, 30, 0.001)
objp = np.zeros((6*7,3), np.float32)
objp[:,:2] = np.mgrid[0:7,0:6].T.reshape(-1,2)
objpoints = []
imgpoints = []
images = glob.glob('./data/chess/*.jpg')
for fname in images:
img = cv2.imread(fname)
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, corners = cv2.findChessboardCorners(gray, (7,6),None)
if ret == True:
objpoints.append(objp)
corners2 = cv2.cornerSubPix(gray,corners,(11,11),(-1,-1),criteria)
imgpoints.append(corners2)
ret, mtx, dist, rvecs, tvecs = cv2.calibrateCamera(objpoints, imgpoints, gray.shape[::-1],None,None)
draw라는 사용자 정의 함수를 만들텐데, 이 함수는 cv2.findChessboardCorners() 함수를 통해 구한 체스판의 코너점과 축의 포인트를 인자로 받아 3차원 축을 그립니다.
def draw(img, corners, imgpts):
corner = tuple(corners[0].ravel())
img = cv2.line(img, corner, tuple(imgpts[0].ravel()), (255,0,0), 5)
img = cv2.line(img, corner, tuple(imgpts[1].ravel()), (0,255,0), 5)
img = cv2.line(img, corner, tuple(imgpts[2].ravel()), (0,0,255), 5)
return img
축의 포인트를 위한 변수를 정의할 것인데, 이 변수는 축을 그리기 위한 3차원 공간 상의 포인트입니다. 길이 3(단위는 체스보드의 크기를 기반으로 함)만큼을 축의 길이로 정합니다. 그래서 X축의 선은 (0,0,0)-(3,0,0)으로, Y축의 선은 (0,0,0)-(0,3,0)으로, Z축은 (0,0,0)-(0,0,-3)인데, 음수인 이유는 카메라의 방향을 나타내기 위함입니다.
axis = np.float32([[3,0,0], [0,3,0], [0,0,-3]]).reshape(-1,3)
자, 이제 각 이미지를 로드하고 이미 상의 체스판의 7×6 그리드를 찾습니다. 만약 발견되면, 이를 Subcorner 픽셀로 정제합니다. cv2.solvePnPRansac() 함수를 사용해 회전과 이동을 계산합니다. 일단 이러한 변환 행렬이 계산되면, 이를 이용해 이미지 평면 상의 축의 포인트들을 투영합니다. 간단히 말해서, 3차원 상의 좌표 (3,0,0), (0,3,0), (0,0,3)에 해당하는 이미지 상의 좌표를 얻는 것입니다. 이 3개의 좌표가 얻어졌다면 앞서 정의한 draw 함수를 통해 그립니다.
for fname in images:
img = cv2.imread(fname)
gray = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY)
ret, corners = cv2.findChessboardCorners(gray, (7,6),None)
if ret == True:
corners2 = cv2.cornerSubPix(gray,corners,(11,11),(-1,-1),criteria)
# Find the rotation and translation vectors.
_, rvecs, tvecs, inliers = cv2.solvePnPRansac(objp, corners2, mtx, dist)
# project 3D points to image plane
imgpts, jac = cv2.projectPoints(axis, rvecs, tvecs, mtx, dist)
img = draw(img, corners2, imgpts)
cv2.imshow('img',img)
k = cv2.waitKey(500)
아래 그림은 위 예제의 실행 시 표시되는 영상 중 하나입니다.
이제, 3차원 축을 파악했으니, draw 함수를 변형해서 3차원 큐브를 이미지에 표시하겠습니다. 수정된 draw 함수는 다음과 같습니다.
def draw(img, corners, imgpts):
imgpts = np.int32(imgpts).reshape(-1,2)
# draw ground floor in green
img = cv2.drawContours(img, [imgpts[:4]],-1,(0,255,0),-3)
# draw pillars in blue color
for i,j in zip(range(4),range(4,8)):
img = cv2.line(img, tuple(imgpts[i]),tuple(imgpts[j]),(255),3)
# draw top layer in red color
img = cv2.drawContours(img, [imgpts[4:]],-1,(0,0,255),3)
return img
변경된 draw 함수에 맞게 기존의 axis 변수도 큐브를 구성하는 8개의 모서리 좌표에 맞게 변경됩니다.
axis = np.float32([[0,0,0], [0,3,0], [3,3,0], [3,0,0],
[0,0,-3],[0,3,-3],[3,3,-3],[3,0,-3]])
실행해보면, 그 결과 중 하나의 이미지는 아래와 같습니다.